1.1.2 CRISPR/Cas9 zur genetischen Modifikation
Im wesentlichen ist der Ablauf, wenn das CRISPR/Cas9 System zur Genmodifikation verwendet wird, der gleiche wie unter 1.1.1 beschrieben. Als Grundlage dient der Streptococcus pyogenes TypII CRISPR Locus. Um dieses CRISPR/Cas System zur genetischen Modifizierung von Säugerzellen verwenden zu können, müssen zunächst alle Komponenten des Systems in die Zellen eingeschleust werden. Vorangegangene Studien haben gezeigt, dass für die Erzeugung von Doppelstrang-Brüchen, die Expression dreier Komponenten ausreicht. Hierbei handelt es sich um die tracrRNA, die pre-crRNA und die Cas9 Nuklease. Dies gilt sowohl in vitro [12,13] als auch in prokaryontischen [14,15] und eukaryontischen Zellen [3,4]. Ein Vektor, der eine Expressionskassette mit den drei Komponenten enthält, kann diese über Transfektion in die Zielzellen einbringen. Für Säugerzellen wird eine Codon-optimierte Cas9 Nuklease genutzt. Zudem wird diese von zwei NLS (nuclear localisation sites) flankiert, so dass eine Lokalisation der Nuklease im Zellkern herbeigeführt wird. Enthält die pre-crRNA Region außerdem eine Restriktionsschnittstelle, kann hier jede beliebige guiding Sequenz eingefügt werden. Die nachfolgenden Abbildungen geben Beispiele für solche Expressionskassetten, in denen die RNAs einzeln oder als crRNA-tracrRNA-Chimär vorliegen können.
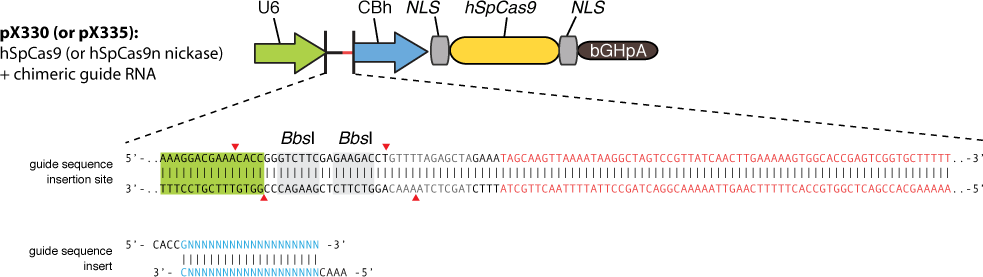
Abb. 6: Aufbau pX330 mit BbsI/BpiI Schnittstelle sowie Aufbau Insert
Ausschnitt aus pX330-U6-Chimeric_BB-CBh-hSpCas9
rote Buchstaben: tracrRNA; blaue Buchstaben: Insert; grün: U6 Promoter; gelb: Cas9 Nuklease [geändert nach 3]
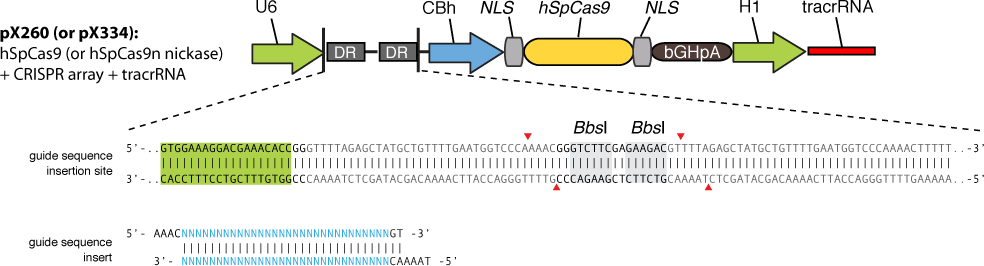
Abb. 7: Aufbau pX260 mit BbsI/BpiI Schnittstelle sowie Aufbau Insert
Ausschnitt aus pX260-U6-DR-BB-DR-Cbh-NLS-hSpCas9-NLS-H1-shorttracr-PGK-puro
blaue Buchstaben: Insert; rot: tracrRNA; grün: U6 Promoter; gelb: Cas9 Nuklease;
DR: directed repeats [geändert nach 3]
Die aufgeführten Abbildungen zeigen Ausschnitte von Plasmiden. Bei der oberen Abbildung handelt es sich um eine solche chimäre guide RNA aus crRNA, tracrRNA und einem synthetischen Verbindungsloop handelt [3,12].
Werden solche Plasmide über Transfektion in Säugerzellen eingebracht, so findet zunächst die Expression des Cas9 Proteins sowie der ein bis zwei RNA Einheiten statt. Danach folgt die Prozessierung der RNA. Der anschließend gebildete Protein-RNA-Komplex interagiert mit der genomischen DNA der Säugerzelle und führt dort zu einem Doppelstrang-Bruch [3,4,16]. Hierbei wird die eigentlich unspezifische Nuklease Cas9, spezifisch zu einer Targetsequenz geleitet. Dies geschieht durch die zuvor in den Bereich der guide RNA klonierte Sequenz. Diese guiding Sequenz ist eine kurze DNA Sequenz des Gens, das modifiziert werden soll. Abhängig vom verwendeten Plasmid, sind solche guiding Sequenzen 20-30 bp lang. Hinter diesen Sequenzen sollte in der genomischen DNA ein PAM (protospacer-adjacent motif) folgen. Im Falle des TypII CRISPR/Cas Systems aus Streptococcus pyogenes setzt sich dieses aus drei Nukleotiden zusammen und lautet NGG [17]. Allerdings wurde auch eine Toleranz gegenüber NAG PAMs nachgewiesen [17,18]. Durchschnittlich kommt ein solches NGG PAM alle 8 Basenpaare vor bzw. alle 4, wenn die NAGs mit einbezogen werden [19,20]. Es kann also davon ausgegangen werden, dass jedes Gen im menschlichen Genom mit Hilfe eines CRISPR/Cas9 Systems modifiziert werden kann. Die gewünschten Doppelstrang-Brüche im betreffenden Gen wiederum führen zur Induktion genetischer Reparaturmechanismen. Dabei handelt es sich um die beiden Mechanismen NHEJ (nonhomologous end joining repair) und HDR (homology directed repair). Werden nun bei der Reparatur der DNA durch diese Mechanismen Fehler eingebracht, so kann dies zu Insertionen oder Deletionen führen [21]. Diese wiederum können eine Verschiebung des Leserasters bedingen und so zu einem dauerhaften Knock-out des Gens führen [22].
Neben der Generierung von Knock-outs, können Varianten des CRISPR/Cas9 Systems für andere Zwecke eingesetzt werden. So können Gene mit Hilfe eines sogenannten CRISPRi (CRISPR interference) Systems, reversible ausgeschaltet werden. Dieses System beinhaltet eine katalytisch inaktive Cas9 Nuklease. Diese kann mit verschiedenen Proteinen, wie etwa Transkriptionsfaktoren, fusioniert werden und so zur Repression oder Aktivierung von Genen führen [23]. Es handelt sich somit um eine Plattform, die zur Entschlüsselung der Funktion bestimmter DNA Regionen genutzt werden könnte [23,24].
Eine weitere Variante ist die Verwendung einer Nickase. Diese ist durch Inaktivierung der RuvC ähnlichen Domäne aus der Cas9 Nuklease hervorgegangen [12,13,14]. Nickasen erzeugen im Gegensatz zu Nukleasen, Einzelstrang-Brüche. Setzt man zwei Nickasen mit nah aneinander liegenden guiding Sequenzen ein, können hierdurch ebenfalls Knock-outs generiert werden. Zudem lässt sich so die Spezifität des gesamten Systems erhöhen [25]. Kurze DNA Stücke können außerdem gezielt ins Genom geschleust werden, wenn diese gleichzeitig mit den Komponenten des CRISPR/Cas9 Systems transfiziert werden [4].
ANHANG – LITERATURVERZEICHNIS
1. Barrangou R, Fremaux C et al.: „CRISPR provides acquired resistance against viruses in prokaryotes.“ Science. 2007 Mar 23;315(5819):1709-12
2. Garneau JE, Dupuis MÈ et al.: „The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA.“ Nature. 2010 Nov 4;468(7320):67-71
3. Cong L, Ran FA et al.: „Multiplex genome engineering using CRISPR/Cas systems.“ Science. 2013 Feb 15;339(6121):819-23
4. Mali P, Yang L et al.: „RNA-guided human genome engineering via Cas9.“ Science. 2013 Feb 15;339(6121):823-6
5. Ishino Y, Shinagawa H et al.: „Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product.“ J Bacteriol. 1987 Dec;169(12):5429-33
6. Makarova KS, Haft DH et al.: „Evolution and classification of the CRISPR-Cas systems.“ Nat Rev Microbiol. 2011 Jun;9(6):467-77
7. Wiedenheft B, Sternberg SH, Doudna JA: „RNA-guided genetic silencing systems in bacteria and archaea.“ Nature. 2012 Feb 15;482(7385):331-8
8. http://crispr.genome-engineering.org/ Datum: 24.02.2014
9. Brouns SJ, Jore MM et al.: „Small CRISPR RNAs guide antiviral defense in prokaryotes.“ Science. 2008 Aug 15;321(5891):960-4
10. Deltcheva E, Chylinski K et al.: „CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III.“ Nature. 2011 Mar 31;471(7340):602-7
11. Semenova E, Jore MM et al.: „Interference by clustered regularly interspaced short palindromic repeat (CRISPR) RNA is governed by a seed sequence.“ Proc Natl Acad Sci U S A. 2011 Jun 21;108(25):10098-103
12. Jinek M, Chylinski K et al.: „A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity.“ Science. 2012 Aug 17;337(6096):816-21
13. Gasiunas G, Barrangou et al.: „Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria.“ Proc Natl Acad Sci U S A. 2012 Sep 25;109(39):E2579-86
14. Sapranauskas R, Gasiunas G et al.: „The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli.“ Nucleic Acids Res. 2011 Nov;39(21):9275-82
15. Magadán AH, Dupuis MÈ et al.: „Cleavage of phage DNA by the Streptococcus thermophilus CRISPR3-Cas system.“ PLoS One. 2012;7(7):e40913
16. Horvath P, Barrangou R: „RNA-guided genome editing à la carte.“ Cell Res. 2013 Jun;23(6):733-4
17. Mojica FJ, Díez-Villaseñor C et al.: „Short motif sequences determine the targets of the prokaryotic CRISPR defence system.“ Microbiology. 2009 Mar;155(Pt 3):733-40
18. Deveau H, Barrangou R et al.: „Phage response to CRISPR-encoded resistance in Streptococcus thermophilus.“ J Bacteriol. 2008 Feb;190(4):1390-40
19. Jiang W, Bikard D et al.: „RNA-guided editing of bacterial genomes using CRISPR-Cas systems.“ Nat Biotechnol. 2013 Mar;31(3):233-9
20. Mali P, Aach J et al.: „CAS9 transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering.“ Nat Biotechnol. 2013 Sep;31(9):833-8
21. Cho SW, Kim S et al.: „Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease.“ Nat Biotechnol. 2013 Mar;31(3):230-2
22. Santiago Y, Chan E et al.: „Targeted gene knockout in mammalian cells by using engineered zinc-finger nucleases.“ Proc Natl Acad Sci U S A. 2008 Apr 15;105(15):5809-14
23. Qi LS, Larson MH et al.: „Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression.“ Cell. 2013 Feb 28;152(5):1173-83
24. Gilbert LA, Larson MH et al.: „CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes.“ Cell. 2013 Jul 18;154(2):442-51
25. Ran FA, Hsu PD et al.: „Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity.“ Cell. 2013 Sep 12;154(6):1380-9
