Für Alle die es nun genauer wissen wollen, hier die Einleitung meiner Masterarbeit mit dem Thema “RNA-guided genetic engineering of mammalian cells via CRISPR/Cas9”.
1.1 CRISPR/Cas System
Das CRISPR/Cas System ist ein aus Bakterien und Archea stammendes System, zur Abwehr von eindringender DNA [1,2]. Es wurde festgestellt, dass Bestandteile dieses Systems genutzt werden können, um das Genom von Säugerzellen gezielt zu manipulieren. So können zum Beispiel Knock‑outs in Säugerzellen hervorrufen können [3,4].
Um die Hintergründe zu verstehen, soll hier nun zunächst die ursprüngliche Funktionsweise des CRISPR/Cas Systems erläutert werden.
1.1.1 CRISPR/Cas in Bakterien und Archea
CRISPR steht für clustered regularly interspaced short palindromic repeats und bezeichnet den Locus im Genom, auf dem alle Komponenten codiert sind. Erstmals wurde CRISPR/Cas in E. coli 1987 beschrieben [5]. Eine dieser zum Knock-out essentiellen Komponenten sind die Cas Nukleasen wobei Cas für CRISPR associated steht. Des weiteren werden nicht codierende RNAs benötigt. Hierzu zählen crRNA (CRISPR derived RNA), welche die sogenannte guiding Sequenz enthält, sowie in einigen Fällen die tracrRNA (trans-acting antisense RNA).
Es gibt drei verschiedene Typen des CRISPR/Cas Systems. Es wird vom TypI bis TypIII gesprochen, die sich vor allem darin unterscheiden, welche Cas Proteine verwendet werden und wie das pre-crRNA Transkript in die kürzeren crRNA Stücke geschnitten und prozessiert wird [6]. Hauptmerkal des TypI ist, dass unter den codierten Cas Genen immer Cas3 ist. Während bei TypII insgesamt nur vier Cas Gene codiert werden, von denen eines immer Cas9 ist. Für die Prozessierung wirde neben der pre-crRNA eine zusätzliche RNA, die tracrRNA, benötigt. TypIII wiederum umfasst die restlichen Systeme und lässt sich in zwei Untergruppen aufteilen [7]. Ein genauerer Blick soll hier auf TypII geworfen werden, da ein solcher Mechanismus für die genetische Modifizierung in Säugerzellen verwendet werden kann. Dies soll am Beispiel des Systems aus Streptococcus pyongenes erfolgen. Selbiges diente nach molekularen Veränderungen, wie der Codon-Optimierung des Cas9 Gens für Säugerzellen sowie Reduzierung der Komponenten auf die essentiellen, als Grundlage der für dieses Projekt genutzten Plasmide [3].
Nachstehende Abbildung zeigt schematisch den CRISPR Locus dieses Bakteriums. Hier sind die vier Cas Gene, die tracrRNA sowie die crRNA, die sich aus Spacern und Wiederholungssequenzen zusammensetzt, lokalisiert. Zur Vereinfachung wird nur Cas9 dargestellt. Die anderen Gene liegen zwischen Cas9 und der Wiederholungssequenz.
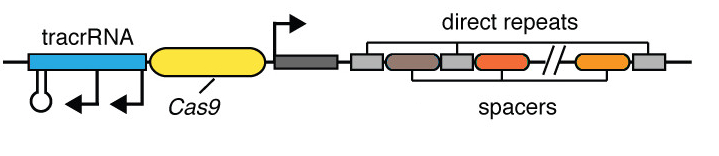
Abb. 1: Schematische Darstellung CRISPR Locus Streptococcus pyongenes [geändert nach 8]
Nach erfolgter Infektion mit Fremd-DNA, kann das weitere Vorgehen in drei Schritte unterteilt werden. Zunächst muss die Fremd-DNA bzw. kurze DNA Fragmente in den CRISPR Locus eingefügt werden. Hierzu wird am vorderen Ende die Wiederholungssequenz dupliziert und als neuer Spacer ein Stück der Fremd-DNA zwischen die repeats eingebaut [2]. Wie dies genau abläuft ist bisher nicht bekannt.

Abb. 2: Schematische Darstellung CRISPR/Cas System DNA Integration [geändert nach 8]
Zunächst erfolgt die Transkription, sowie die Prozessierung der crRNA. Hierbei wird die komplette Region mit Wiederholungssequenzen und Spacern transkribiert, so dass eine pre-crRNA entsteht.

Abb. 3: Schematische Darstellung CRISPR/Cas System Transkription [geändert nach 8]
Die pre-crRNA wird anschließend mit Hilfe der RNAseIII zunächst in kürzere Stücke geschnitten [9]. Für diesen Schritt wird zudem die tracrRNA benötigt, welche mit einem zur Wiederholungssequenz komplementären Bereich an die pre-crRNA bindet. Nach dem Schneiden wird das 5′ Ende dieses crRNA-tracrRNA Duplex prozessiert und kann nun zusammen mit dem Cas9 Protein einen Komplex formen [10].

Abb. 4: Schematische Darstellung CRISPR/Cas System Reifung crRNA und Komplex Bildung [geändert nach 8]
Der entstandene Komplex interagiert als letzter Schritt nun mit der Fremd-DNA. Dies geschieht mit Hilfe der guiding DNA, die den Komplex zur Targetsequenz führt und mit dieser binden kann [11]. Cas9 ist eine unspezifische Nuklease, die zwei aktive Zentren besitzt. Die HNH Domäne schneidet den zur guiding Sequenz komplementären Strang, während die RuvC ähnliche Domäne den nicht komplementären Strang schneidet [12]. Durch das Schneiden beider Stränge wird ein Doppelstrang-Bruch erzeugt. Hierdurch kann die Fremd-DNA inaktiviert und abschließend mit Hilfe endogener Mechanismen abgebaut werden. Hiermit schließt die Abwehr eindringender DNA durch das CRISPR/Cas System ab.

Abb. 5: Schematische Darstellung CRISPR/Cas System Interaktion mit genomischer DNA [geändert nach 8]
Anhang – Literaturverzeichnis
1. Barrangou R, Fremaux C et al.: „CRISPR provides acquired resistance against viruses in prokaryotes.“ Science. 2007 Mar 23;315(5819):1709-12
2. Garneau JE, Dupuis MÈ et al.: „The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA.“ Nature. 2010 Nov 4;468(7320):67-71
3. Cong L, Ran FA et al.: „Multiplex genome engineering using CRISPR/Cas systems.“ Science. 2013 Feb 15;339(6121):819-23
4. Mali P, Yang L et al.: „RNA-guided human genome engineering via Cas9.“ Science. 2013 Feb 15;339(6121):823-6
5. Ishino Y, Shinagawa H et al.: „Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product.“ J Bacteriol. 1987 Dec;169(12):5429-33
6. Makarova KS, Haft DH et al.: „Evolution and classification of the CRISPR-Cas systems.“ Nat Rev Microbiol. 2011 Jun;9(6):467-77
7. Wiedenheft B, Sternberg SH, Doudna JA: „RNA-guided genetic silencing systems in bacteria and archaea.“ Nature. 2012 Feb 15;482(7385):331-8
8. http://crispr.genome-engineering.org/ Datum: 24.02.2014
9. Brouns SJ, Jore MM et al.: „Small CRISPR RNAs guide antiviral defense in prokaryotes.“ Science. 2008 Aug 15;321(5891):960-4
10. Deltcheva E, Chylinski K et al.: „CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III.“ Nature. 2011 Mar 31;471(7340):602-7
11. Semenova E, Jore MM et al.: „Interference by clustered regularly interspaced short palindromic repeat (CRISPR) RNA is governed by a seed sequence.“ Proc Natl Acad Sci U S A. 2011 Jun 21;108(25):10098-103
12. Jinek M, Chylinski K et al.: „A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity.“ Science. 2012 Aug 17;337(6096):816-21
